近年来随着双特异性抗体和CAR-T等新兴治疗手段的出现,弥漫大B细胞淋巴瘤(DLBCL)患者的生存得到了显著提高。尽管如此,仍有部分患者一线R-CHOP标准治疗后面临复发或难治(R/R)。更好地理解R/R DLBCL的潜在机制有助于早期识别潜在进展或复发风险高危的患者。为此,中国和瑞典的研究者合作开展了一项基于2805例R-CHOP治疗DLBCL患者的回顾性研究,发现TP53、MYD88、SPEN和MYC基因与预后差相关。综合基因组学和转录组学结果,建立了一个基于24个基因表达水平的预测模型,可作为DLBCL诊断2年内进展或复发的独立预测模型,且对不同细胞起源和分子学分型均具有临床预测价值,并开发了在线评分工具(https://lymphprog.serve.scilifelab.se/app/lymphprog)。【肿瘤资讯】将相关内容进行了整理,以飨读者。
研究背景
DLBCL是最常见的侵袭性淋巴瘤,约占非霍奇金淋巴瘤(NHL)的30%。大多数患者一线R-CHOP标准治疗可获得长期缓解,约1/3的患者一线治疗难治或最终复发,多在诊断几年内复发。R/R DLBCL预后差,早期识别诊断后几年内的潜在高危复发难治人群对改善这部分DLCBL患者的预后至关重要。
国际预后指数(IPI)是临床最为常用的DLBCL危险分层评分,但IPI评分未将多项临床指标、生物标志物、基因表达谱以及免疫组化或分子学检测手段等纳入其中。为进一步阐明药物耐药和疾病进展的相关机制,建立识别早期R/R DLBCL危险因素的分子学模型,开展了本项研究。分析887例诊断后2年内疾病进展或复发的DLBCL患者的临床特征、突变谱和基因表达谱,并与同期诊断2年仍处于缓解状态的1918例患者进行对比,致力于建立可有效预测DLBCL早期复发进展风险的预后模型工具。
研究方法
该研究包括7个DLBCL队列,队列1(本研究队列)包括2001年至2015年瑞典或中国诊断并接受R-CHOP方案治疗的161例DLBCL,其中73例为瑞典患者,88例为中国患者。另外6个队列为已发表且可获得临床/DNA突变/基因表达谱数据的DLBCL队列。
研究结果
本研究队列161例患者中50例(31%)2年内疾病进展或复发。对比分析显示,预后差患者的临床特征为年龄大、ABC亚型、疾病进展期和IPI评分更高。已发表队列的2644例患者临床特征分析显示,32%的患者2年内疾病进展或复发,R/R患者的高危特征与本研究队列大致相符。此外,各研究队列的PFS未见统计学显著差异。
进一步分析2290例DLBCL肿瘤的突变谱并识别出99个可进一步分析的基因,包括KMT2D、PIM1、TP53、BCL2、HIST1H1E、MYD88和CREBBP在内的24个基因突变发生率≥10%。对预后差的702例患者的突变谱进行分析,20个发生率≥10%。预后差患者的常见突变基因包括KMT2D(32.8%)、PIM1(27.4%)、TP53(26.9%)、BCL2(21.8%)、MYD88(19.9%)、TMSB4X(18.2%)、HIST1H1E(18.0%)和BTG2(15.7%)。对比发现,预后差患者中4个基因(TP53、MYD88、SPEN和MYC)突变显著富集(q<0.1)、8个基因(CD83、BCL6、SGK1、ACTB、TMEM30A、CARD11、P2RY8和TNFRSF14)显著缺失。Cox回归分析显示上述4个富集基因与PFS缩短相关,而8个缺失基因与PFS延长相关。4个富集基因(TP53+MYD88+SPEN+MYC)中的任一基因均较单个基因HR更高(1.55 vs 1.26~1.46)。约53%的患者可按LymphGen亚型分类,预后差组A53和MCD亚型更常见,而EZB和ST2亚型在预后良好组占比更多,表明不同的突变谱与DLBCL早期进展或复发相关。
为进一步比较预后差和预后良好组的基因表达谱,分析了RNA测序数据库327例患者的差异表达基因(DEGs),其中122例归类为预后差组。预后差组的DEGs包含一小部分上调基因(n=73,BCL2、BLNK、CXorf21、CD72、IGLL5、CCND2等)和一大批下调基因(n=1472),下调基因中的49个为淋巴瘤中的常见突变基因和免疫检查点相关基因(CTLA4、TIGIT和CD80)。DEGs中,4个基因(SGK1、CD83、BCL6和P2RY8)在预后差DLBCL中突变更为少见,而SPEN基因突变更常见。进一步分析微阵列芯片数据库的DEGs(n=1049,其中336例被归类为预后差组),并将其与RNA测序数据库进行对比。结果显示,372个为重合基因,占相应队列DEGs的24%和39%。超过98%的重合DEGs与两队列的上调或下调一致,表明不同数据库和不同患者人群的整体一致性。
对RNA测序数据库和微阵列芯片数据库进行整合,并将样本随机分为发现队列和验证队列(分别占70%和30%)。采用单变量Cox回归模型分析PFS和基因表达水平的相关性,共识别出656个预后基因,其中242个基因与372个DEGs重合。输入发现队列964例患者242个基因的表达水平和每例患者的2年结局,在1000个组合中,一个包含24个基因在内的模型被选为最佳风险预测模型。其多个基因在相关细胞间通路中发挥重要作用,包括BCR信号通路(RFTN1)、蛋白激酶通路(SGK1)、凋亡通路(FAS)、细胞分化标记(CD28、CD3E、ITGAX、LY75、TNFRSF13C/BAFFR)、细胞因子和生长因子(PLAU)和转录因子(LMO2、EGR1、TCF7)。计算每个肿瘤的24个基因表达评分,根据最佳分界值,将患者分为高危和低危组。高危组PFS显著缩短,更多患者在2年内疾病进展或复发。
该模型对2年结局的阳性预测值(PPV)为0.61,阴性预测值(NPV)为0.79,整体精确性为74%。多因素分析显示该预测模型为2年结局关键临床因素调整后的独立预测因素(p<0.001),且ROC峰值分析超越了COO亚型、关键临床特征、多种突变状态对2年结局的预测价值。验证队列(n=412)中的分析结果与发现队列一致,2年结局的PPV、NPV和整体精确性分别为0.55、0.75和70%。在有双打击或双表达数据的患者中,该模型仍具有独立预测价值,且预测意义最佳。
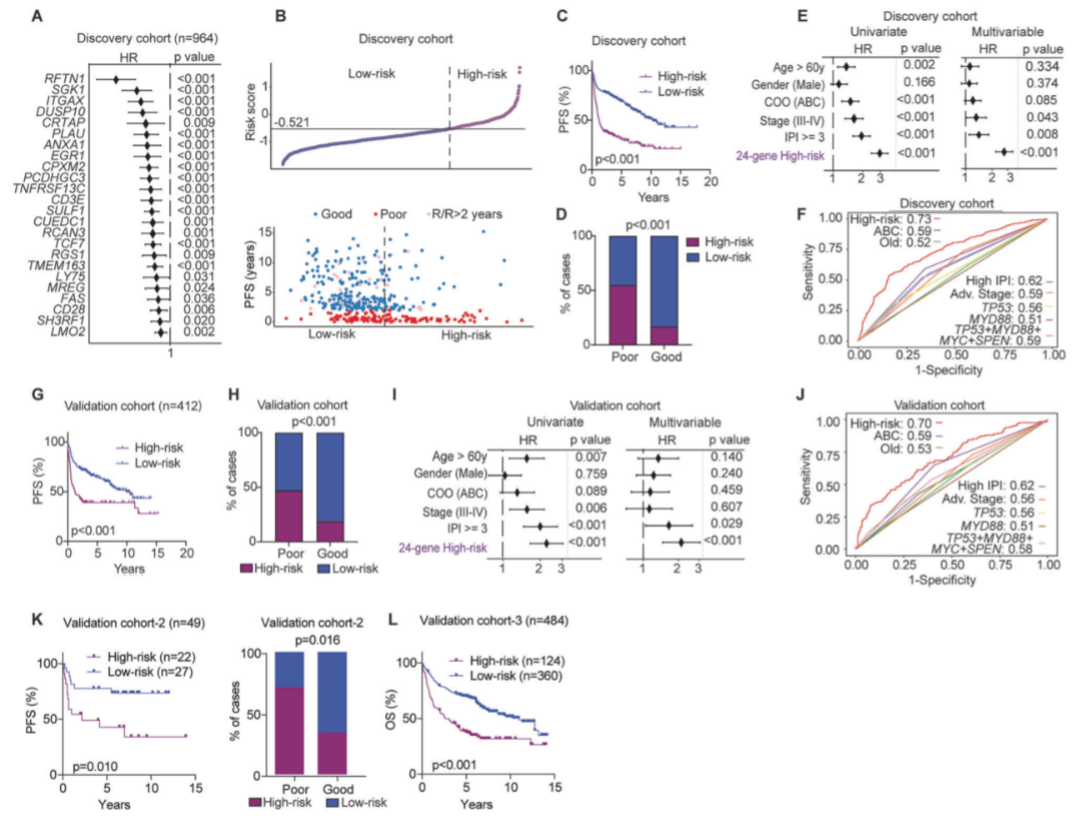
图1 预测DLBCL治疗结局的独立基因表达印记的发现和验证过程
分析24个基因表达评分与COO亚型的相关性,ABC亚型高危患者占比更多。与低危组相比,ABC和GCB亚型中的高危患者PFS均显著缩短。且在COO无法分类患者中,该风险预测模型仍可识别出高危患者。分析24个基因表达评分与DLBCL分子学亚型的相关性,该风险预测模型仍可在不同LymphGen亚型中以及不能归类亚型中仍具有预测价值。
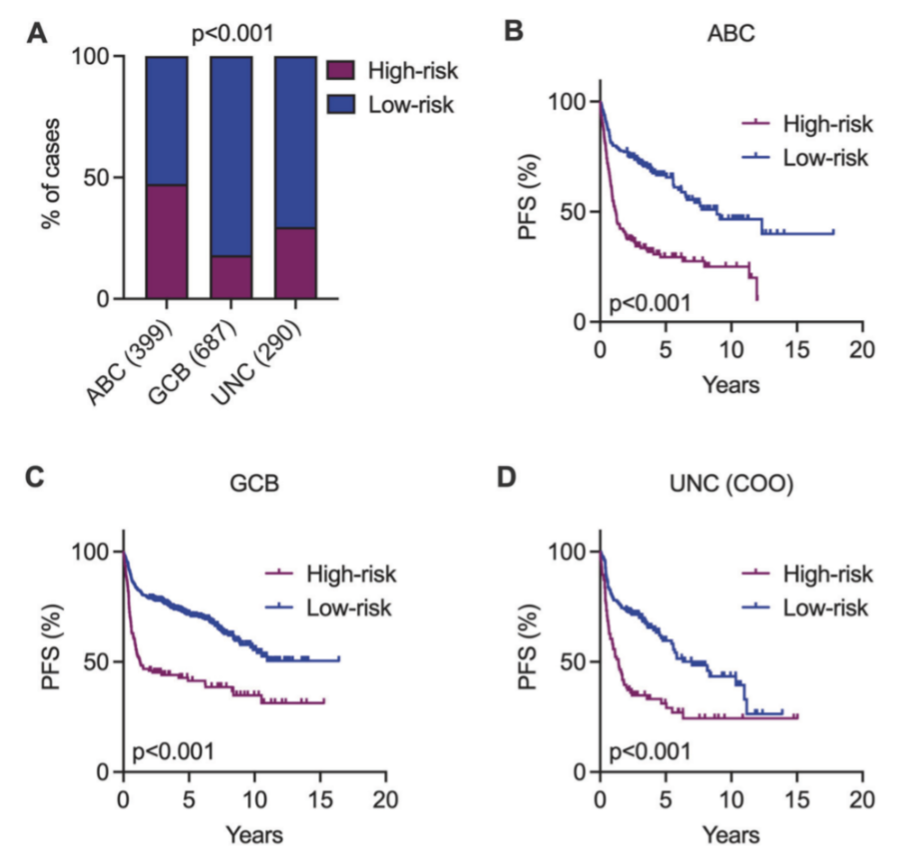
图2 24个基因表达评分在不同细胞起源DLBCL中的独立分层价值
研究结论
整合RNA测序和微阵列芯片结果的24个基因表达评分模型是DLBCL诊断后2年内疾病进展或复发的独立预测模型,基于此模型识别出的高危患者2年内更容易发生疾病进展或复发,且该模型在不同COO亚型和不同分子学分型中仍具预测价值。
研究者说
近年来基因组学和转录组学研究以及单细胞测序在DLBCL中进行得热火朝天,但对早期疾病进展或复发的影响和预测因素仍知之甚少。该研究共包括7个队列,涵盖2800多例接受R-CHOP标准治疗的DLBCL,建立了一个新的可独立预测早期疾病进展的评分模型,且在不同细胞起源、不同分子学亚型中均具有预测价值。对于密切监测R-CHOP治疗后的临床结局,识别高危诊断后2年内进展或复发的DLBCL患者,并及时进行临床干预意义重大。
简而言之,对于DLBCL患者而言,包含24个基因表达水平的风险预测模型可识别2年内进展或复发的高危患者,针对这部分患者,临床或需要采用标准治疗与新兴疗法相结合的加强治疗手段,例如双特异性抗体、CAR-T等,以进一步提高缓解深度,延长高危患者生存。
小结与展望
随着测序技术的飞速发展和应用,DLBCL新的基因分型不断被提出,对预测生存临床意义重大。约1/3的DLBCL最终疾病进展和复发,多发生在诊断后1-2年。早期识别高危进展或复发风险患者,加强临床干预对延长这部分患者生存结局尤为重要。新的预测早期疾病进展风险模型的提出,有助于临床识别早期复发进展患者,提供更加个性化的治疗方案,以进一步延长生存。
随着对DLBCL发生和发展进制更深入的理解和治疗策略的不断优化,新的早期复发预测模型在DLBCL中的应用前景广大,有望早期识别高危DLBCL患者,以改善临床结局。
Weicheng Ren, Hui Wan, Sulaf Abd Own, et al. Genetic and transcriptomic analyses of diffuse large B-cell lymphoma patients with poor outcomes within two years of diagnosis [J]. Leukemia, 2023.
排版编辑:肿瘤资讯-Amiee











 苏公网安备32059002004080号
苏公网安备32059002004080号


