乳腺上皮的体细胞拷贝数异常(CNAs)发生率及其在肿瘤发生发展中的作用还不清楚。近日,Nature Genetics发表一项研究,使用单细胞DNA测序方法在BRCA1/2突变或野生型正常乳腺上皮细胞中探索了CNAs的发生情况和生物学特征[1],为CNAs在肿瘤基因组中的作用提供重要依据。【肿瘤资讯】整理如下,以飨读者。

研究背景
体细胞突变通过逐步累积可导致肿瘤的发生,拷贝数变异(CNA)是肿瘤中常见的基因剂量突变,尤其在乳腺癌中,CNAs经常引发重要的驱动事件,如 ERBB2 的扩增和 PTEN 的丧失,这些变异也常是基因组不稳定性肿瘤(如乳腺癌)中转录变异的主要来源。研究表明,导管原位癌(DCIS)中存在广泛的染色体或染色体片段的复制或丢失,导致CNA和结构变异(SVs),这一现象与浸润性乳腺癌相似[2]。癌前不典型导管增生(ADH)同样存在广泛的CNA突变[3],这些研究表明CNAs在乳腺癌进化的早期阶段就已发生。然而,正常乳腺上皮细胞中最早的CNA发生率、进化时机及其分布特征尚不明确。
由于CNA的发生率较低,全面测定其特征面临一定挑战。为解决这一问题,研究团队开发了一种大规模单细胞全基因组测序(scWGS)技术,可以有效检测单个细胞中的CNAs。本研究利用这一技术,探索了 BRCA1/2 胚系突变携带者与BRCA野生型正常乳腺上皮组织中的CNA突变发生率、染色体分布及谱系特异性。
研究结果
正常乳腺上皮细胞CNAs的发生率与细胞类型相关
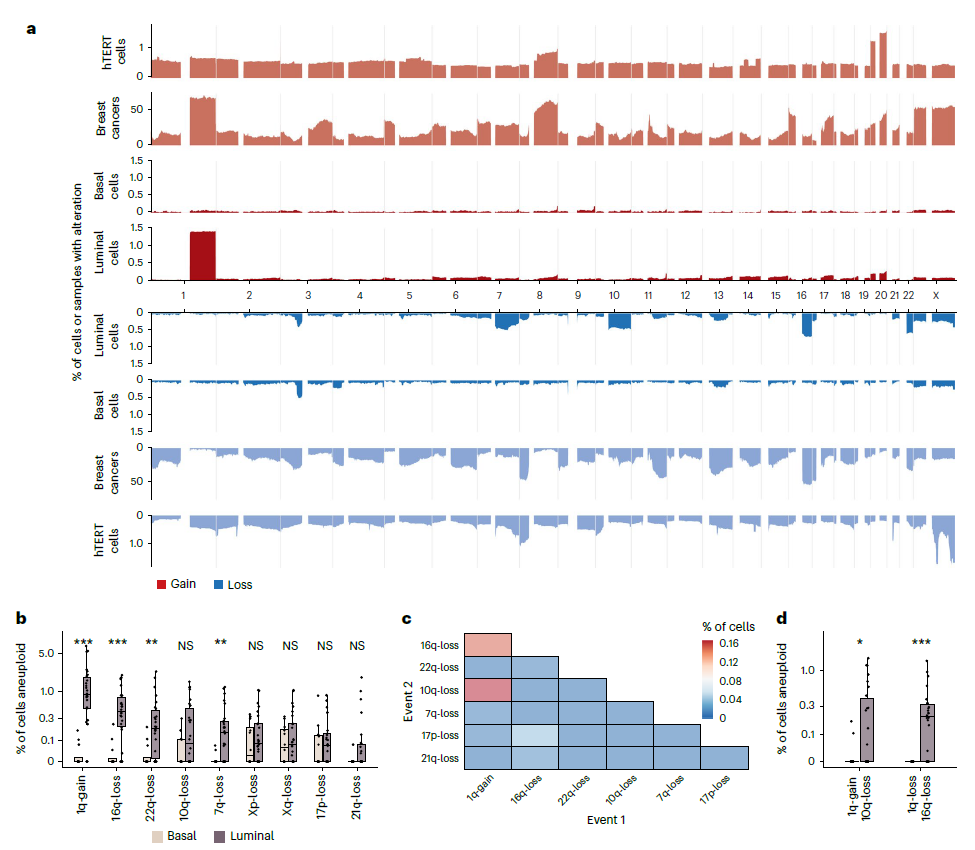
研究收集了携带胚系致病性 BRCA1(n=12)或 BRCA2(n=7)突变以及 BRCA1/2 野生型(n=9)女性的乳腺组织。通过单细胞分解技术,组织样本被分为腔面型细胞和基底细胞群体。使用单细胞测序分析,共覆盖28例供者,分析了49,238个单细胞基因组(图1a)。在全基因组拷贝数分析中,二倍体基因组和非整倍体基因组的范例分别展示在图1b和1c中。
非整倍体细胞的定义为至少一个染色体臂的获得或丢失,尽管发生率较低,但在每个样本中均有观察到。大约 3.25% 的细胞表现为1至4个非整倍体染色体臂(单非整倍体)。特定变异如1q增益和16q、10q、22q及7q丢失在不同供者间较为常见(图1d-g)。在腔面型细胞中,非整倍体细胞的出现率显著高于基底细胞(3.73% vs 1.38%,P=0.001,图1h)。且携带 BRCA 突变的个体相比野生型个体非整倍体细胞的发生率略有升高,分别为 BRCA1 突变(3.63%)、BRCA2 突变(3.65%)和野生型(2.45%)(P=0.13和0.11,图1i)。多变量分析表明,腔面型细胞与非整倍体细胞的增加密切相关(P=0.0002)。

腔面型细胞中的重复性CNAs与乳腺癌相似
腔面型细胞与基底细胞表现出不同的CNA分布。多次出现的CNAs主要集中在腔面型细胞中(图2a),其中包括1q增益(腔面型细胞为1.53% vs 基底型细胞为0.03%,P=0.00002)、16q丢失(0.61% vs 0.03%,P=0.00011)、22q丢失(0.39% vs 0.03%,P=0.0022)、7q丢失(0.26% vs 0.01%,P=0.0011)和10q丢失(0.31% vs 0.07%,P=0.083,图2b)。尽管X染色体丢失在腔面型和基底型细胞中的发生率相似(0.20% vs 0.11%,P=0.58),腔面型细胞中这些CNAs的重复性较强。
与555例乳腺癌全基因组测序数据对比发现,部分在腔面细胞中常见的基因事件,如1q增益和16q、22q丢失,也在晚期乳腺癌中较为常见(图2a)。尽管7q丢失在正常乳腺上皮数据中较为常见,但在乳腺癌样本中却相对罕见(图2a)。
为确定复发性CNAs是否可由潜在突变偏倚解释,对比了本研究结果和13,569例野生型永生化乳腺组织细胞系的单细胞基因组。和正常乳腺上皮scWGS不同,这一细胞系的CNAs分布在整个基因组中相对均匀(图2a)。异常发生率和特征也和近来一项显示错分离率受核染色体位置影响的研究不同[4]。提示正常乳腺上皮染色体中较高的CNAs发生率是组织和细胞类型特异性过程,可能和谱系分化和/或上皮细胞定向相关。
在具有超过1个非整倍体染色体臂的细胞中,最常见的联合变异是1q增益/16q丢失和1q增益/10q丢失(图2c)。这些联合变异主要集中在腔面型细胞中,平均频率分别为0.29%和0.27%。这一发现与近期研究结果一致,后者表明1q增益/16q丢失的克隆可能是癌症诊断的前驱,并且在癌症诊断前十余年即可出现[5]。
等位基因特异性分析揭示多个独立CNAs事件
通过对15个具有大量多倍体细胞的样本进行SIGNALS分析,研究发现不同染色体臂的等位基因分布表现出强烈的不平衡聚集。例如,样本B2-23中,既有包含所有常见CNAs的细胞,也有部分细胞表现为1q增益/10q丢失和1q增益/16q丢失(图3a)。等位基因特异性拷贝数分析表明,这些事件是至少两次独立获得的,提示多倍体人群不是单个克隆的扩增,而是多个独立的异常事件(图3c)。

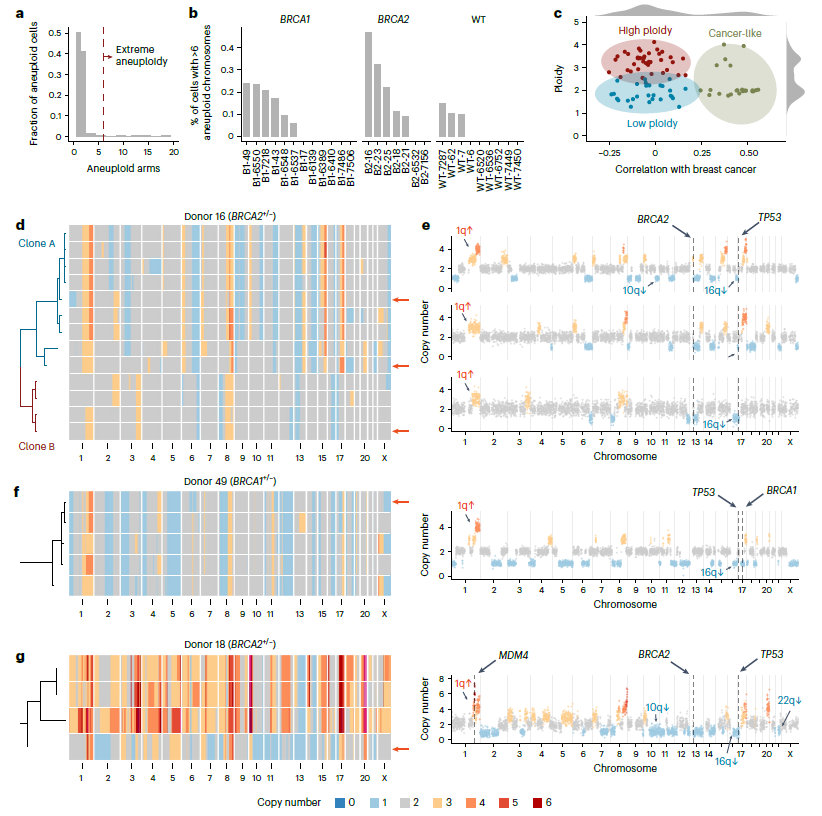
极端非整倍体细胞罕见但是存在
尽管大多数非整倍体细胞仅含有1到2个CNAs,研究仍发现少数细胞具有多个CNAs(图4a)。在极端非整倍体细胞中(即超过6个非整倍体染色体臂的细胞),其发生率较低,仅为0.1%(图4b)。将这些细胞与乳腺癌的特征进行对比,研究发现22个细胞与乳腺癌基因组相似,并将其标记为“癌样”基因组(图4c)。这些“癌样”细胞均来自3个高危供者样本,并表现出 BRCA1 或 BRCA2 1拷贝的丢失,同时17p区域(包含TP53)的一个等位基因丢失,提示P53功能的丧失。这些细胞显示出强烈的克隆特征,并提示它们可能是乳腺癌早期的演化克隆(图4d-g)。
研究讨论
这项乳腺上皮的大规模单细胞基因组分析显示正常组织的多种体细胞CNAs特征。研究发现,所有上皮细胞中约有 3.25% 的细胞表现为非整倍体(包括染色体臂的获得或丢失)。在腔面型细胞和基底型细胞中,CNA的发生率分别为 3.73% 和 1.38%。CNAs主要集中在腔面型细胞中,特征性变异包括 1q 增益、以及 10q、16q、22q 和 7q 的丢失。该CNAs特征具有细胞谱系和染色体特异性,可能反映了组织特异性的演变过程。因此,数据提示,CNAs是正常乳腺组织上皮细胞体细胞突变谱的重要组成部分,且这些变异呈现出明显的细胞类型和染色体特异性。
研究还发现,正常乳腺上皮细胞的CNAs特征与浸润性乳腺癌的全基因组拷贝数特征高度相似。例如,腔面型细胞中的 1q增益 和 16q丢失 常常共存,这一共存的变异在低级别导管原位癌(DCIS)和腔面A型乳腺癌中通常是唯一的异常。数据支持这一现象可能是早期的肿瘤发生事件,并且这些变异可通过多个独立的等位基因事件形成。1q增益 和 16q丢失 可能通过不平衡易位事件(如 der(1;16) )发生,导致染色体臂的融合。近期研究表明,der(1;16) 是乳腺癌初期的关键基因异常,并且这些克隆会随着时间积累额外突变,最终发展为浸润性癌症[6]。这些发现强调了 1q增益 和 16q丢失 在癌前阶段的重要性,靶向这些早期变异可能成为乳腺癌预防或监测的新策略。
部分CNAs变异可能与乳腺癌的亚型和预后相关。例如,10q丢失 与 PTEN 基因的丧失密切相关,PTEN缺失在基底型乳腺肿瘤中尤为常见。PTEN 的丢失通常发生在 BRCA1 LOH(失去杂合性)之前[7],并提示BRCA1/2单倍剂量不足可能导致染色体不稳定性,进而促进非整倍体的发生[8]。在BRCA1/2突变携带者中,非整倍体细胞的发生率呈增加趋势,这进一步支持BRCA1/2单倍剂量不足可能是CNAs获得的一个重要原因。
研究还观察到,部分 BRCA1/2 突变携带者的细胞表现出广泛的CNAs,这些细胞的特征类似于BRCA突变相关癌症。大多数这些细胞携带 1q 和 10q 或 16q 的CNAs,并且可能是通过丧失野生型BRCA等位基因形成的。
研究结论
综上所述,该研究发现腔面型乳腺上皮细胞中的CNAs发生率高于基底型细胞,并且BRCA突变携带者在腔面型细胞中的CNAs异常具有增加的趋势。这些腔面型CNAs的发生模式与乳腺癌中的CNAs特征相似,提示它们可能是肿瘤发生的早期标志。未来的研究应进一步探索这些谱系特异性突变及选择机制与肿瘤形成之间的关系,特别是如何利用这些早期变异进行癌症预防和监测。
[1] Williams MJ, et al. Luminal breast epithelial cells of BRCA1 or BRCA2 mutation carriers and noncarriers harbor common breast cancer copy number alterations. Nat Genet. 2024 Nov 20.
[2] Wang K, et al. Archival single-cell genomics reveals persistent subclones during DCIS progression. Cell. 2023;186(18):3968-3982.e15.
[3] Simpson PT, et al. Molecular evolution of breast cancer. J Pathol. 2005;205(2):248-54.
[4] Sjoerd JK, et al. Nuclear chromosome locations dictate segregation error frequencies. Nature. 2022,607:604-609.
[5] Nishimura T, et al. Evolutionary histories of breast cancer and related clones. Nature. 2023;620(7974):607-614.
[6] Cross WC, et al. New paradigms in clonal evolution: punctuated equilibrium in cancer. J Pathol. 2016;240(2):126-36.
[7] Martins FC, et al. Evolutionary pathways in BRCA1-associated breast tumors. Cancer Discov. 2012;2(6):503-11.
[8] Sedic M, et al. Haploinsufficiency for BRCA1 leads to cell-type-specific genomic instability and premature senescence. Nat Commun. 2015:6:7505.
声明:材料由阿斯利康支持,仅供医疗卫生专业人士参考
审批编号:CN-149537
过期日期:2025-12-09
排版编辑:肿瘤资讯-展思懿











 苏公网安备32059002004080号
苏公网安备32059002004080号


