肿瘤细胞的一个主要特征是表观遗传调控元件的异常激活,引起重塑肿瘤转录组,最终促进癌症的进展和耐药。有关卵巢癌(OC)的致病机制尚不十分明确,近期,来自中国的一项研究发现,卵巢癌特异高表达的转录因子KLF5可促进卵巢癌增殖和转移过程,并通过重塑同源重组关键基因RAD51的转录,促进卵巢癌PARP抑制剂耐药[1]。【肿瘤资讯】特对研究主要内容进行梳理,以飨大家。
研究背景
卵巢癌(OC)是死亡率最高的妇科恶性肿瘤之一[2],上皮性卵巢癌(EOC)约占OC患者的90%,包括高级别浆液性卵巢癌 (HGSOC)、低级别浆液性卵巢癌(LGSOC)、卵巢子宫内膜样癌(OEC)、卵巢透明细胞癌(OCCC)和黏液性卵巢癌(MOC)等亚型[3]。除了常见的TP53突变和BRCA1/2、ARID1A、CDK2等蛋白质编码基因中少数罕见但重要的复发性体细胞突变外,OC的总体突变率非常低,这使得针对蛋白质编码基因突变的药物开发极具挑战性[4]。
卵巢癌的标准一线治疗包括减瘤手术和铂类化疗。然而,近70%的晚期OC患者会出现铂耐药,导致肿瘤复发,生存期明显缩短[5]。因此,迫切需要在其他领域进行研究,例如探索基因组的非编码区和肿瘤转录组,以了解OC进展的潜在机制并制定新的治疗策略。肿瘤转录组的多样性和复杂性是癌症的主要特征,表观遗传调控元件,如启动子、增强子和超级增强子(SE),在肿瘤中出现特异性或异常激活,通过重塑肿瘤转录组产生肿瘤成瘾或肿瘤特异性转录本(TST)来促进肿瘤发生、转移和耐药。PARP抑制剂的开发在治疗患者方面显示出强大的效果,不过,耐药是接受PARP抑制剂治疗的OC患者的常见问题,OC是否通过表观遗传调控元件的异常激活来控制转录组重塑并调节PARP抑制剂耐药性,目前尚未有过报道。
研究结果
1.OC 中SE异常激活
研究者在OC中发现了2481个异常激活的SE元件。为揭示不同OC亚型中SE相关的关键信号通路,研究者分析了SE富集的邻近基因,发现OEC亚型富集铁死亡、NF-κB等信号通路富集;HGSOC亚型趋化性和胆碱代谢信号通路富集;OCCC亚型主要富集于TGF-β和Wnt信号通路。所有三种亚型均在Hippo、MAPK和转录异常信号通路中富集。总的来说,研究结果提供了对OC中异常激活的SE及其在不同OC亚型中的存在和特征的全面理解。

图1 描述OC中的超级增强元件
(A) 用于识别OC中异常激活的SE的分析策略的流程图。(B) 维恩图显示了每个OC亚型中SE的数量。(C-E) OVCAR5中SE的数量、igrpv1、ES2细胞和相邻代表性基因的表示。(F) 矩阵显示了在不同细胞类型中检测到的SE的成对相似性。(G) 三种OC亚型中SE邻近基因富集的特定和共享的KEGG信号通路
2.SE驱动KLF5转录和KLF5自转录调控
火山图显示,KLF5、PAX8、SOX17、ELF3和HOXB3在OC组织中高表达。此外,研究者构建了这些TF的潜在下游靶基因的调控网络,在OVCA420和OVCAR5 OC细胞中观察到KLF5启动子上游的两个SE调控元件被激活。在OVCA420细胞中,两个SE显示H3K4me1和H3K27Ac的组蛋白修饰信号,与EP300和BRD4结合,H3K4me3的修饰信号水平较低。研究者还观察到,KLF5和BRD4蛋白结合发生在它们的启动子区域,这表明KLF5可能受到SE的转录调控和自我调控。
研究者使用enCRISPRi系统抑制这两种SE的活性,发现单独或共同抑制两种SE的活性,能显著下调H3K27Ac水平和KLF5 mRNA水平。同样,抑制两种SE活性可以抑制OVCA420细胞的集落形成和侵袭能力。此外,研究者通过ChIP-qPCR实验证实了KLF5和BRD4在KLF5启动子区域的结合,并在OVCA420细胞中设计了靶向KLF5和BRD4的小干扰RNA(siRNA),敲低KLF5或BRD4显著抑制KLF5启动子活性。BRD4敲低显著降低了KLF5 mRNA和蛋白质水平。综上所述,这些结果提示SE异常激活调节KLF5和KLF5的转录,并且KLF5进一步结合其自身的启动子区域以维持自身高转录表达。
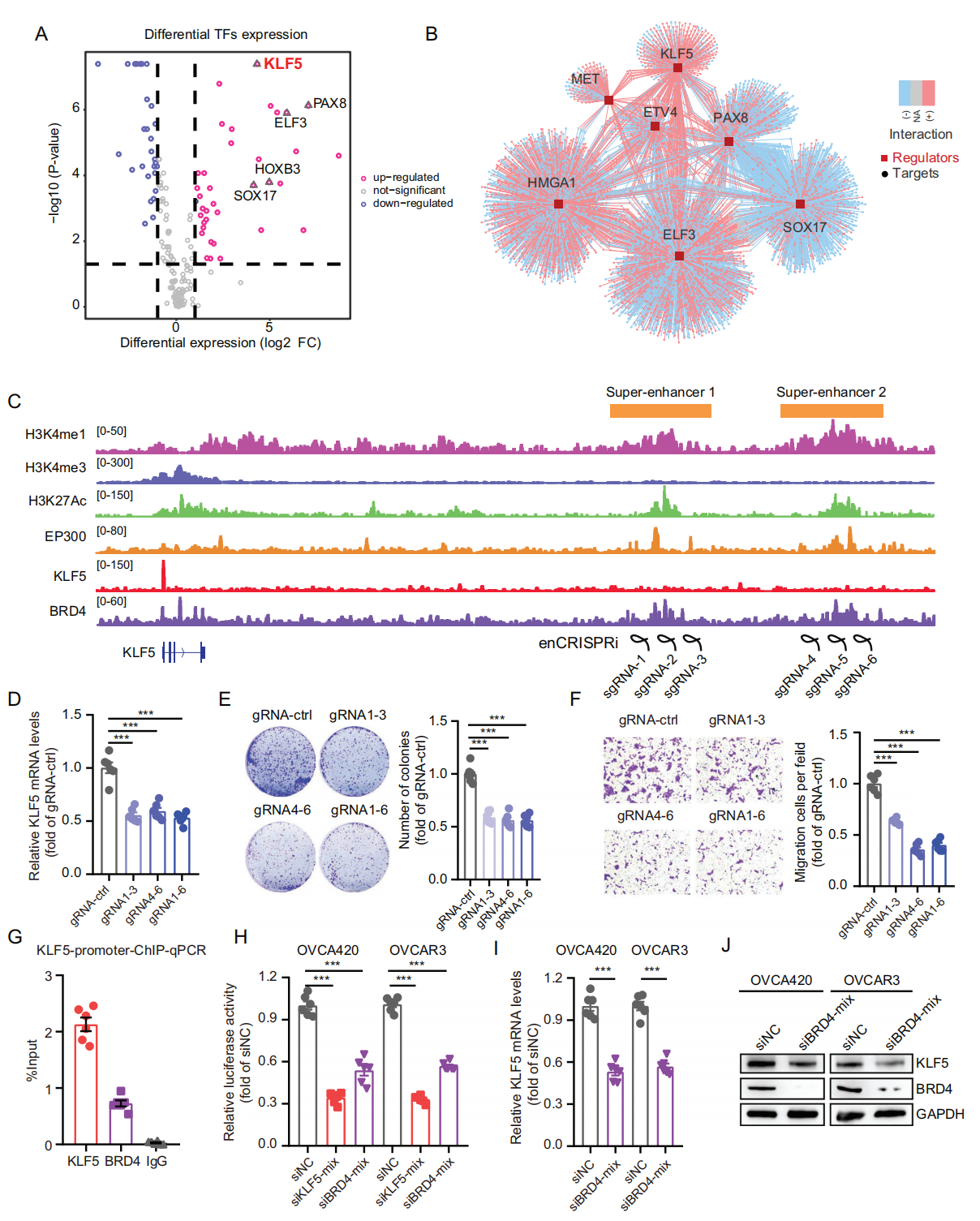
图2 SE在OC中驱动KLF5转录和KLF5自转录调节
(A) 使用 FUSCC 队列 RNA-seq 数据,火山图显示 OC 中 TF 的差异表达,包括 8 个正常卵巢组织和 30 个 OC 组织。(B) TF 的差异表达及其下游基因的调控网络。(C) H3K4me1、H3K4me3、H3K27Ac、EP300、KLF5和BRD4在KLF5启动子和两个上游 SE 处的 ChIP-seq。(D) 转染 dCas9-KRAB、靶向KLF5 SE 的 sgRNA 和 MCP-LSD1 慢病毒的 OVCA420 细胞中KLF5的 mRNA 水平。(E-F) 使用 enCRISPRi 慢病毒阻断KLF5 SE 可抑制 OVCA420 细胞中的集落数量形成和迁移能力 (G) ChIP-qPCR 测定显示OVCA420 细胞中KLF5和BRD4在KLF5启动子处的结合。(H) OVCA420和OVCAR3细胞中用KLF5或BRD4 siRNA转染的KLF5启动子的荧光素酶活性。(I-J) BRD4 sirna转染的KLF5在OVCA420和OVCAR3细胞中的mRNA水平和蛋白水平
3. OC特异性KLF5高表达,与不良预后相关
研究者发现KLF5在OC、胆管癌、胃癌等肿瘤中高表达。其中,KLF5在OC中的表达差异尤为显著。在FUSCC队列1的OC组织中发现了类似的KLF5高表达模式,KLF5高表达的OC患者总生存率和无病生存率较低。研究者在KLF5FUSCC队列2中验证了KLF5蛋白水平,发现KLF5蛋白水平在OC组织中也高表达。KLF5蛋白水平高的 OC 患者的总生存率和无病生存率也较低。

图3 KLF5在 OC 中特异性过度表达,并且在临床上与患者预后相关
(A)TCGA 数据集中不同正常和肿瘤样本中KLF5的 mRNA 水平。(B) FUSCC 队列 1 中正常和 OC 样本中KLF5的 mRNA 水平。(C-D) FUSCC 队列 1 中总生存和无病生存的 Kaplan-Meier 曲线。(E) FUSCC 队列 2 的 74 个正常组织和 165 个 OC 组织中KLF5的代表性免疫染色图像。(F) 正常组织和 OC 组织中KLF5蛋白水平的免疫印迹。(G-H) 用KLF5蛋白水平分层的FUSCC队列2的总生存期和无病生存期的Kaplan-Meier曲线
4.KLF5驱动OC的生长和转移
研究者发现KLF5的敲低显著抑制了OVCA420和TOV21G OC细胞中的集落形成和迁移。KLF5高表达与细胞增殖之间存在显著的正相关性,表明KLF5在促进OC细胞增殖过程中起着至关重要的作用。研究者还发现,KLF5基因敲除可显著降低OC细胞的肿瘤大小,OC细胞中KLF5敲低可减少SUV摄取和并减少腹部肿瘤结节数量。

图4 KLF5驱动 OC 肿瘤生长和转移
(A-B) 用KLF5 siRNA 或对照 siRNA转染的 OVCA420 和 TOV21G 细胞的克隆形成测定和迁移测定。(C) Depmap 数据集中 OC 细胞系中KLF5 mRNA 水平与 CRISPR 影响值(增殖能力)之间的相关性。(D) 感染 Cas9 和KLF5 sgRNA 的 OVCA420 和 SKOV3 细胞中KLF5蛋白水平的免疫印迹。(E) CCK8测定。(F-G)克隆形成实验和迁移实验。(H) 裸鼠的异种移植瘤。(I) KLF5的敲除减少了异种移植肿瘤的重量。(J) PET-CT显示感染Cas9和KLF5 sgRNA的TOV21G细胞的腹部肿瘤转移。(K-L) 感染Cas9和KLF5 sgRNA的TOV21G细胞腹膜腔的相对SUV最大值和结节数
5.KLF5调控RAD51转录和HRR通路
KLF5敲低细胞中下调的前5个信号通路富集于DNA复制、错配修复、剪接体、HR和细胞周期。研究者在OVCA420细胞中对KLF5进行了ChIP-seq,获得了2497个KLF5结合位点,主要位于基因启动子区域,32个靶基因可能受KLF5转录调控,32个基因中有18个与细胞增殖相关。研究者发现抑制KLF5会减少RAD51灶点并增加γ-H2AX 灶点,表明KLF5在调节卵巢癌细胞的HRR中发挥着至关重要的作用。

图5 KLF5调节RAD51转录和HRR通路
(A) 在用KLF5 siRNA 或对照 siRNA转染的OVCA420细胞中,前六个富集的 KEGG 下调通路和三个上调的通路。(B) GSEA富集了转染KLF5 siRNA或对照siRNA的OVCA420细胞的细胞周期和同源重组通路。(C) KLF5在OVCA420细胞中的全基因组结合位点,大部分位于启动子区域。(D) KLF5 ChIP-seq和RNA-seq数据的韦恩图显示了32个在OVCA420细胞中受KLF5转录调控的基因。(E) Depmap 数据集中的 OVCA420 中的KLF5靶基因表明这些基因参与了 OC 细胞增殖。(F) OVCA420中KLF5的 ChIP-seq 数据显示KLF5在RAD51启动子区域的结合。(G) 用KLF5 siRNA或对照siRNA转染的OVCA420、SKOV3和TOV21G细胞中RAD51、p-H2AX、KLF5蛋白水平的免疫印迹。(H) 用KLF5 siRNA 或对照 siRNA转染的 OVCA420 和 TOV21G 细胞中的彗星测定。(I)免疫荧光染色。(J-K) RAD51灶和γ-H2AX灶的定量结果
6.KLF5与EHF、ELF3形成转录复合物
研究者发现,EHF和ELF3在基因组中可能与KLF5重叠。KLF5敲低抑制了EHF和ELF3蛋白水平,表明KLF5可能是最关键的调节蛋白。研究者观察到KLF5与ELF3启动子以及上游SE区和EHF启动子区结合,表明KLF5也可能调节EHF和ELF3的转录。
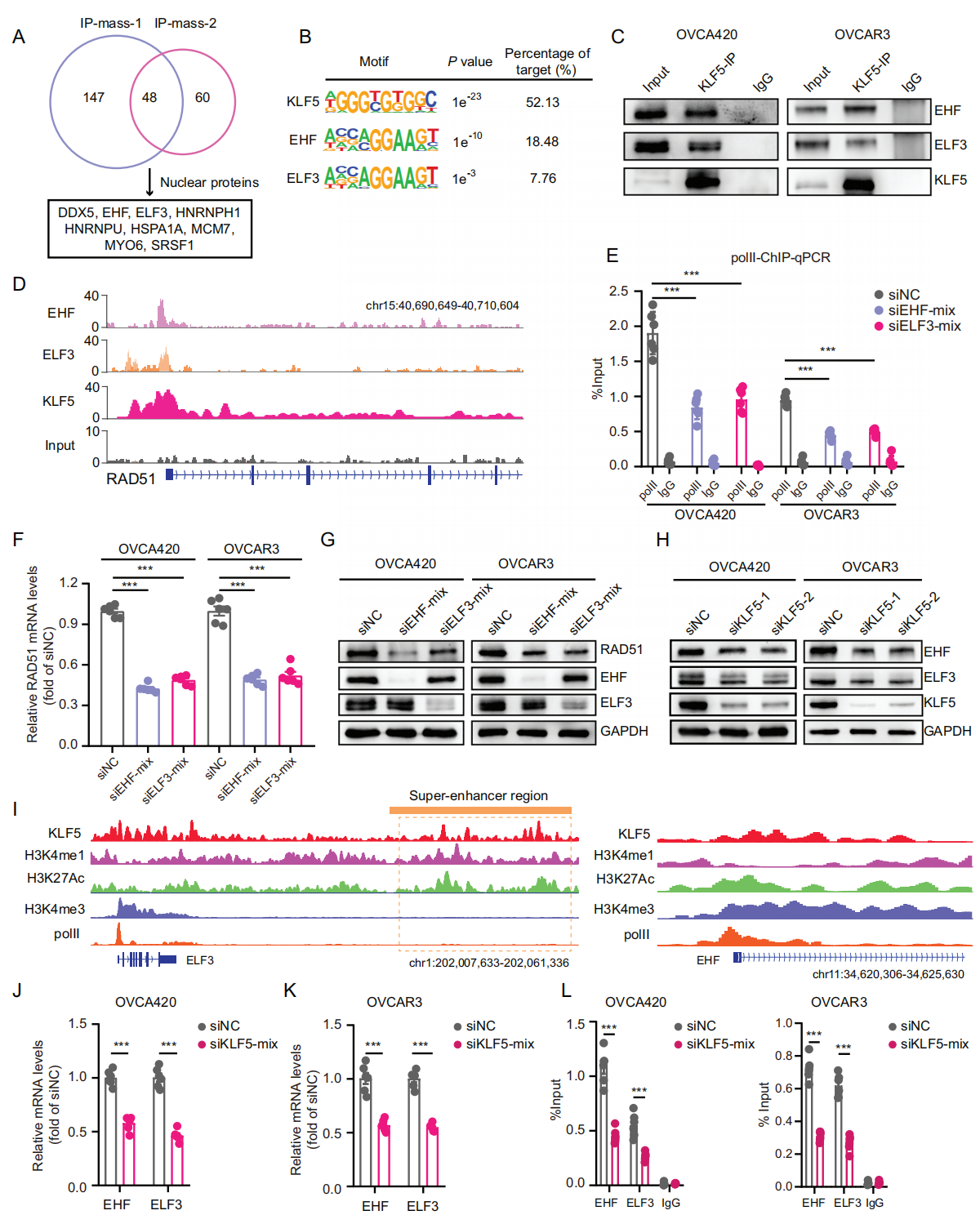
图6 KLF5与EHF和ELF3形成转录复合物
(A) 免疫沉淀-质谱法。(B) OVCA420 细胞中KLF5、EHF和ELF3的基序分析。(C) 免疫共沉淀分析显示OVCA420 和 OVCAR3 细胞中内源性KLF5、EHF和ELF3之间的相互作用。(D) ChIP-seq 数据显示EHF、ELF3和KLF5在RAD51中富集OVCA420 细胞中的启动子区域。(E) 在转染EHF、ELF3 siRNA或对照siRNA的OVCA420和OVCAR3细胞中,polII在RAD51启动子区域的富集能力。(F-G) 用EHF、ELF3 siRNA或对照siRNA转染的OVCA420和OVCAR3细胞中的RAD51 mRNA水平和蛋白质水平。(H) 用KLF5 siRNA或对照siRNA转染的OVCA420和OVCAR3细胞中的EHF、ELF3和KLF5蛋白水平的免疫印迹。(I) KLF5、H3K4me1、H3K27Ac、H3K4me3和polII的ChIP-seq数据显示,在OVCA420细胞中,KLF5富集于ELF3启动子区、SE区和EHF启动子区。(J-K) 用KLF5 siRNA 或对照 siRNA转染的 OVCA420 (J) 和 OVCAR3 (K) 细胞中的EHF和ELF3 mRNA 水平。(L) 用KLF5 siRNA或对照siRNA转染的OVCA420细胞和OVCAR3细胞中RAD51启动子区域中EHF和ELF3的富集能力
7.EHF和ELF3高表达并与不良预后相关
EHF蛋白高表达的OC患者表现出较低的总体生存率,但与无病生存率没有显著相关性。ELF3蛋白高表达的OC患者表现出较低的总生存率和无病生存率。KLF5、EHF和ELF3蛋白水平较高的OC患者表现出较低的总生存率和无病生存率。以上数据表明,KLF5联合EHF和ELF3可能是OC患者的有效预后因素。
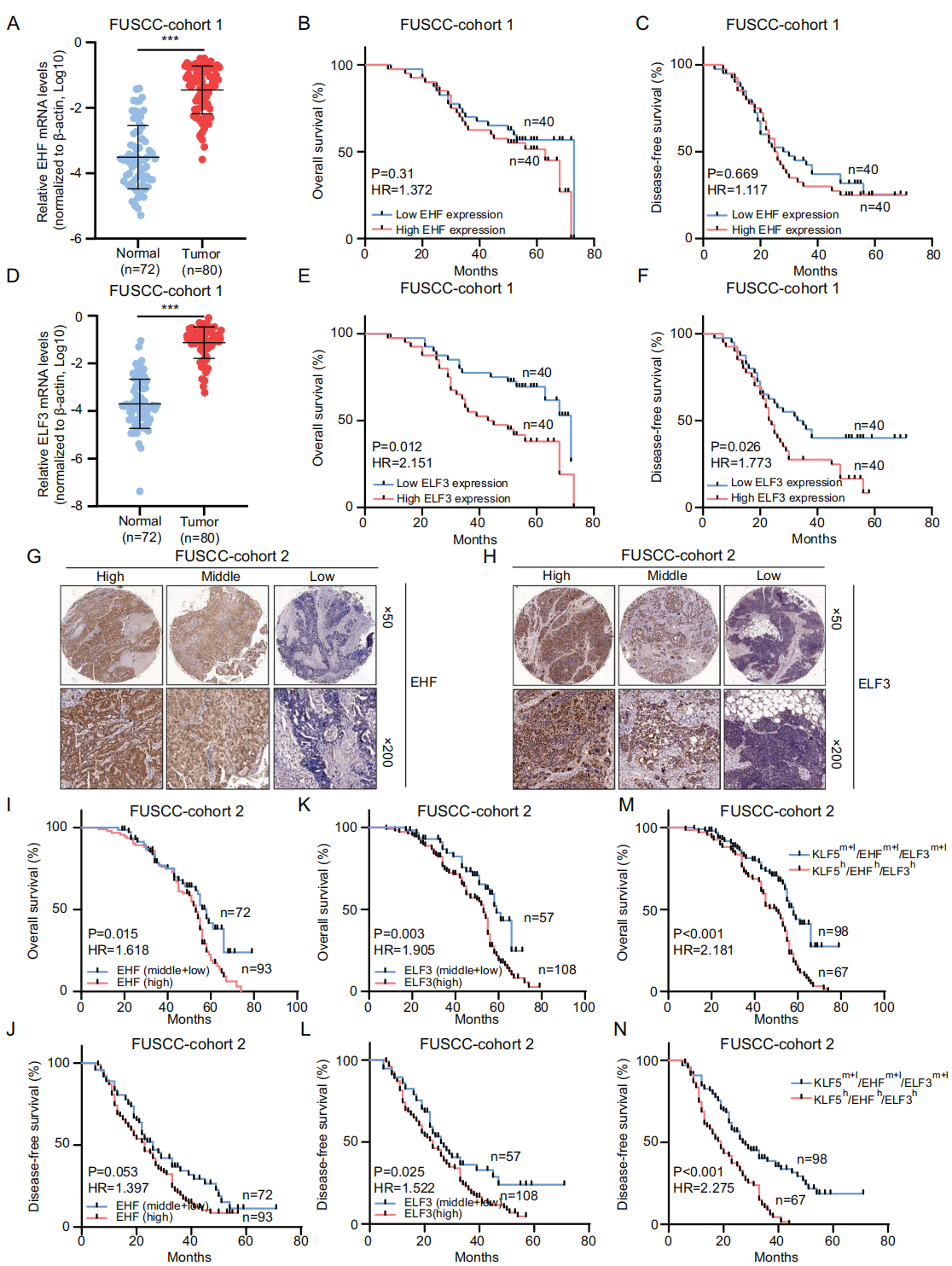
图7 EHF和ELF3在OC患者中的临床意义
(A) FUSCC 队列 1 中正常样本和 OC 样本中的 EHF mRNA 水平。(B-C) FUSCC 队列 1 中总生存期和无病生存期的 Kaplan-Meier 曲线。(D) FUSCC 队列 1 中正常和 OC 样本中ELF3的 mRNA 水平。(E-F) FUSCC 队列 1 中总生存和无病生存的 Kaplan-Meier 曲线。(G-H)免疫染色图像。(I-N) Kaplan-Meier 曲线
8.靶向KLF5增加OC患者对PARP抑制剂耐药的敏感性
PARP抑制剂耐药OC患者表现出KLF5 SE的异常激活,并促进KLF5高表达。KLF5通过重塑RAD51转录和增强HRR途径来调控OC中的奥拉帕利耐药性。
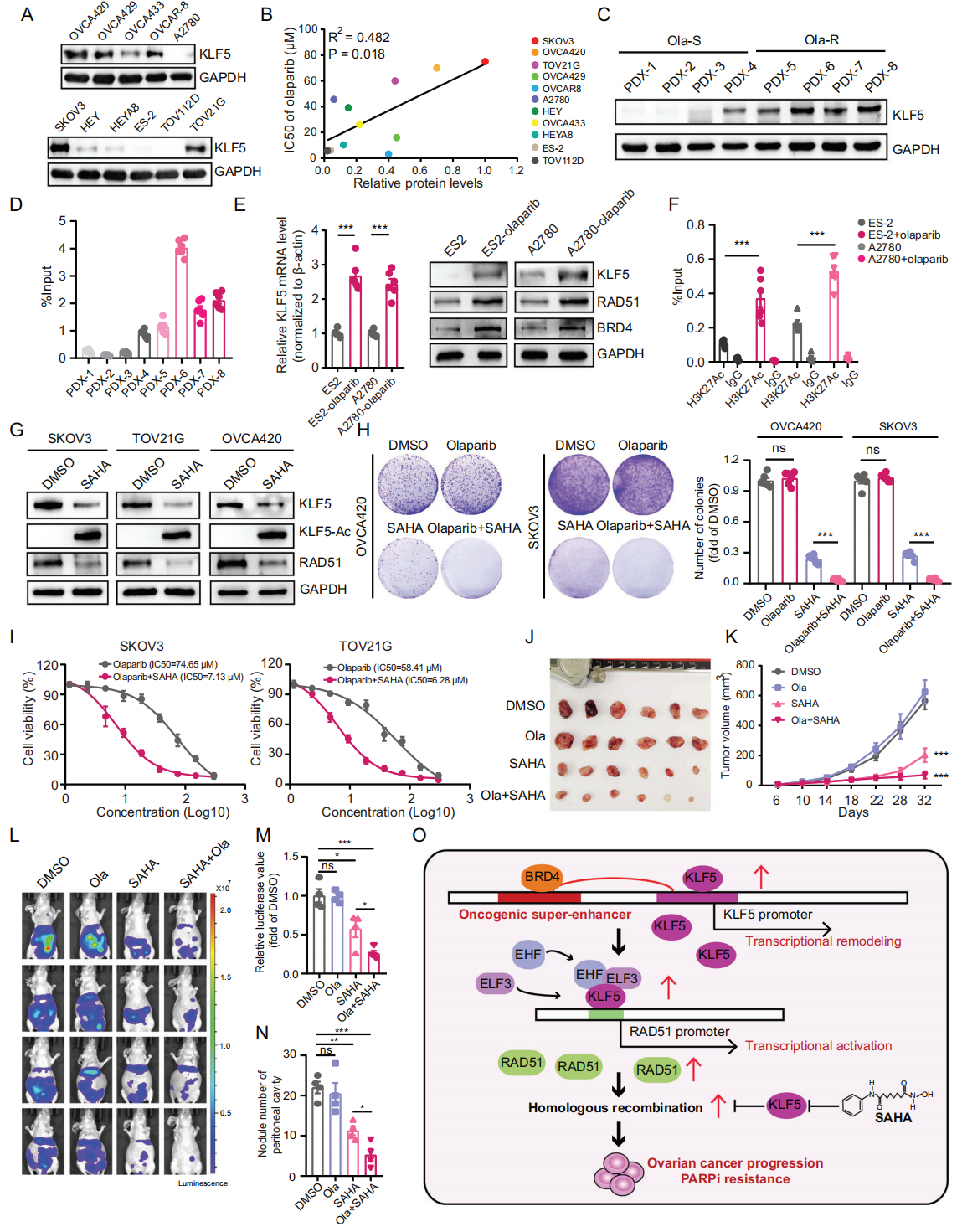
图8 KLF5调控 OC 对 PARP 抑制剂的敏感性
(A) 11 种 OC 细胞系中KLF5蛋白水平的免疫印迹。(B) OC细胞系中KLF5蛋白水平与olaparibIC50值之间的相关性。(C) OC olaparib敏感 (Ola-S) 或耐药 (Ola-R) PDX 样本中KLF5蛋白水平的免疫印迹。(D) 通过 H3K27Ac 信号的 ChIP-qPCR 测定olaparib敏感或耐药的 OC PDX 样品中KLF5超级增强子 2 的活性。(E) ES2、A2780 和olaparib处理的 ES2 和 A2780 细胞中的KLF5 mRNA 水平(左)和KLF5、RAD51、BRD4蛋白水平(右)。(F) KLF5超级增强子2的活性通过 H3K27Ac 信号的 ChIP-qPCR 测定 ES2、A2780 和olaparib处理的 ES2 和 A2780 细胞中的 H3K27Ac 信号。(G) 用 DMSO 或 SAHA (1µm)处理的 SKOV3、TOV21G 和 OVCA420 细胞中KLF5和RAD51蛋白水平的免疫印迹。(H) 克隆形成实验结果。(I) olaparib或olaparib联合SAHA处理SKOV3和TOV21G细胞的IC50值。(J-K) 异种移植物肿瘤和肿瘤体积。(L-N) 荧光素酶图像、荧光素酶值、腹腔结节数显示在BALB/ c裸鼠中,DMSO、olaparib、SAHA、olaparib联合SAHA处理TOV21G细胞的腹腔肿瘤转移。(O) SE驱动KLF5的示意图
研究结论
该研究鉴定了OC中异常激活的SE调控元件,发现SE驱动转录因子KLF5在OC患者和PARP抑制剂耐药患者中的相对特异性表达。KLF5表达与OC患者预后不良相关,并可在体内和体外驱动肿瘤进展,是OC进展和PARP抑制剂耐药的关键调控因子。
[1] Wu Y, Chen S, Shao Y, et al. KLF5 Promotes Tumor Progression and Parp Inhibitor Resistance in Ovarian Cancer. Adv Sci (Weinh). 2023;10(31):e2304638.
[2] Siegel RL, Miller KD, Wagle NS, Jemal A. Cancer statistics, 2023. CA Cancer J Clin. 2023;73(1):17-48.
[3] Ciucci A, Buttarelli M, Fagotti A, Scambia G, Gallo D. Preclinical models of epithelial ovarian cancer: practical considerations and challenges for a meaningful application. Cell Mol Life Sci. 2022;79(7):364.
[4] Macintyre G, Goranova TE, De Silva D, et al. Copy number signatures and mutational processes in ovarian carcinoma. Nat Genet. 2018;50(9):1262-1270.
[5] Lheureux S, Gourley C, Vergote I, Oza AM. Epithelial ovarian cancer. Lancet. 2019;393(10177):1240-1253.
声明:材料由阿斯利康支持,仅供医疗卫生专业人士参考
审核编号:CN-134628
排版编辑:肿瘤资讯-PP











 苏公网安备32059002004080号
苏公网安备32059002004080号


