诸如残留肿瘤、肿瘤分级以及国际妇产科联盟(FIGO)评分等临床病理参数常用于预测卵巢癌患者的存活情况,然而高级别浆液性卵巢癌(HGSOC)患者的5年存活率依旧维持在30%左右。因此,为了持续寻求改进HGSOC预后评估工具,本研究提出了一种创新的基于基因表达的分子预后评分系统(mPS)。该评分系统是基于一个新颖的20基因标志,通过最小绝对收缩和选择算子(LASSO)与Cox回归分析方法得出,其在预测预后方面具有良好表现。
背景介绍
根据组织学类型和分级,上皮性卵巢癌(EOC)被划分为不同的类别。尽管通过细胞减灭手术和基于铂的化疗取得了初步反应,但HGSOC仍然占据了EOC相关病例的70%,并且在初次诊断后的10年内有超过75%的死亡率。这可能是由于HGSOC内部高度的肿瘤遗传异质性和染色体不稳定性,导致化疗或治疗抵抗。因此,寻找有效的基因标志物或预后标记是HGSOC迫切未满足的临床需求。
生存预测考虑了多种因素,如年龄、FIGO分期、组织学、残留病变和肿瘤复发。然而,基于这些传统临床信息的预测在产生稳健的预后方法方面的潜力有限。这是因为各种分子以及免疫因素之间复杂的相互作用导致HGSOC内部反应不一。最近,基于转录组轮廓的HGSOCs的分子亚型已被识别。使用各种聚类算法最常见的共识亚型包括间充质型、免疫反应型、分化型和增殖型。尽管这些分子亚型在组间显示出明显不同的生物学途径调控,但使用癌症基因组图谱(TCGA)HGSOC队列数据时,对患者生存的影响相对较小。先前已有报道,基因标志物可能在确定癌症患者的生存方面发挥潜在且重要的作用,包括卵巢癌。使用101个预后基因标志物预测HGSOC生存的方法已被应用。在各种癌症中,如乳腺癌、结肠癌和前列腺癌,已研究或报告了使用分子基因标志物作为预后标记的方法。
在此,我们提出开发一种mPS,这是一种基于仅20个预测基因的表达和从最小绝对收缩和选择算子(LASSO)-Cox回归得出的关联系数来对HGSOCs的预后进行分层的机器学习方法。图1中展示了所提出的研究设计。
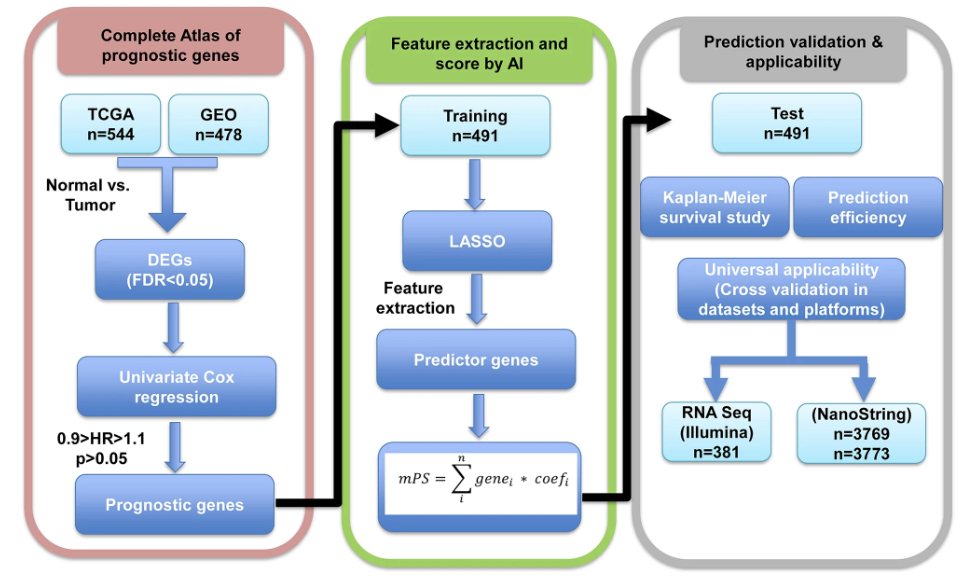
在本研究中,我们考虑了1022名受试者/样本,并筛选或仅考虑了在TCGA和基因表达综合数据库(GEO)中常见的10,225个基因。这里使用了相同或类似平台(Affymetrix人类U133A微阵列或Affymetrix人类基因组U133 plus 2.0阵列)的基于微阵列的表达分析来筛选后续分析的共同基因。这些跨不同数据集的共同基因进一步筛选,以获得基于Cox(比例风险)回归模型的HGSOC的预后基因标志。最后,通过在HGSOC的训练数据集上应用LASSO-Cox回归模型,进一步修剪预后基因和特征提取。这导致获得预测标记以及随后用于获得mPS的衍生系数,最终确定测试或验证数据集中的预后(图1)。
研究方法
本研究从TCGA和GEO数据库下载了原始微阵列数据集,包括TCGA-OV和多个GSE系列数据集。通过RMA方法对数据进行预处理和标准化,使用相关性矩阵排除异常值。利用R和Bioconductor的limma包进行差异基因表达分析,筛选出具有显著差异表达的基因。通过单变量Cox比例风险回归分析,筛选出影响HGSOC患者生存的基因。进一步采用LASSO-Cox回归模型进行多变量分析,构建分子预后评分(mPS)。使用Kaplan-Meier生存曲线和ROC曲线评估mPS的预测能力。通过GO和KEGG数据库进行基因富集分析,揭示与HGSOC相关的生物学途径。数据分析使用R/Rstudio完成。
研究结果
1.卵巢癌与正常卵巢组织之间的差异基因表达和基因集富集分析(GSEA)
我们挑选了至少具有0.7最小基因表达矩阵相关性的10,225个基因和1,016个样本组成的数据子集。本研究的数据涵盖了方法学章节所述的五个不同数据集。包括988个卵巢癌样本和28个代表无卵巢肿瘤迹象的卵巢表面上皮细胞的正常样本。
为了观察样本基因表达轮廓间的距离,我们构建了多维尺度图。利用前500个变异最大的基因计算样本间的成对距离,揭示了样本的分离或聚类模式。值得注意的是,同一数据集中的样本倾向于聚集在一起,而非根据样本类型(正常对癌症)进行聚类,这表明在进一步分析前需要去除批次效应。这一发现强调了在深入分析前解决批次效应的重要性。为了减少由不同数据集引起的批次效应,我们实施了相应的校正步骤。此调整旨在建立一个统一的log2表达比率模式,确保不同数据来源的受试者和样本之间的一致性。这一过程为后续分析提供了更加坚实和一致的基础,有助于更精确地探究基因表达的变化。
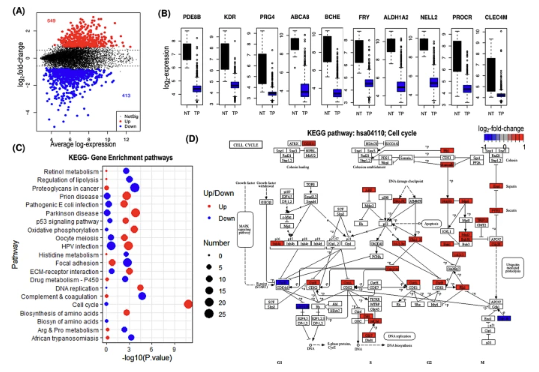
在正常(n=28)和原发性HGSOC(n=973)样本间进行的差异基因表达分析显示,与无肿瘤迹象的卵巢表面上皮组织的正常样本相比,有649个基因在原发性HGSOC肿瘤中表达下调,而473个基因表达上调。基于调整后的p值(FDR<0.05)和倍数变化,图2B展示了最显著的十个失调基因。特别值得注意的是,上调的基因包括CP(铜蓝蛋白铁氧化酶)、FOLR1、TOP2A、CRABP2、MAL、SOX17、CKS2、TPX2、S100A2和UBE2C,而下调的基因顶部是ABCA8、ALDH1A2、BCHE、EFEMP1、NELL2、HBB、TCEAL2、SFRP1、HBA2和FLRT2。
为揭示HGSOC中涉及的生物途径,对1,122个差异表达基因(包括649个下调和473个上调基因)进行了基因富集分析。根据基因本体(GO)数据库的分类,上调基因主要富集在细胞周期过程、细胞周期转换、细胞/核分裂、染色质组织、染色体分离和DNA复制等生物学过程中。KEGG途径分析揭示了细胞周期、补体和凝血级联、DNA复制、氧化磷酸化、ECM-受体相互作用以及药物代谢-细胞色素P450等途径的调控异常(图2C)。
考虑到细胞周期相关分子在肿瘤中的关键作用,我们对细胞周期途径进行了深入分析。利用Bioconductor的‘Pathview’工具集,将原发肿瘤的差异表达基因映射到细胞周期途径(hsa04110)上。在25个差异上调的分子中,有23个与HGSOC中的细胞增殖和肿瘤增长显著相关。这些细胞周期相关分子包括ARF、Ink4a(CDKN2A)、CycD、CycA、CycB、Cdc7、ChK1、MCM(小染色体维持蛋白复合体组分)等(图2D)。
2.风险模型构建
在本研究中,通过与正常样本的比较,共鉴定出1,062个在肿瘤中差异表达的基因,这些基因的校正p值(FDR)小于0.05(附录A补充表S2)。这些基因经过单变量Cox回归分析,并通过log-rank检验(p值<0.05)以及限定风险比(HR)大于1.1或小于0.9进一步筛选。最终,共筛选出122个基因,其中63个与HGSOC患者的预后恶化相关(HR>1.1, p值<0.05),59个与预后改善相关(HR<0.9, p值<0.05)
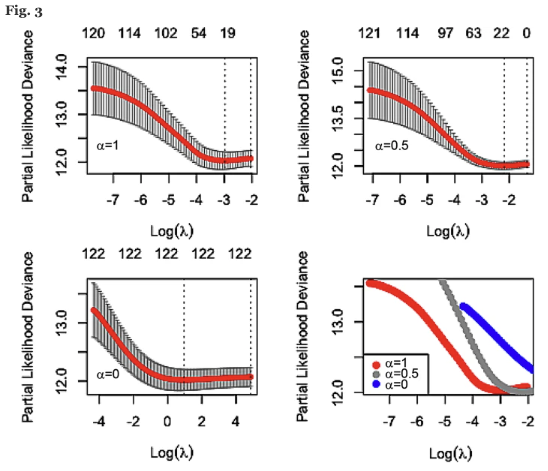
这122个筛选出的基因随后被用于构建LASSO估计模型,该模型基于来自TCGA和GEO数据队列的491个随机选择的样本组成的训练数据集。生成了一个log(Λ)与部分似然偏差图,通过不同的alpha(α)值,发现当α=1时模型拟合最优,这表明了LASSO回归模型的适用性(图3)。在后续分析中,采用10折交叉验证,同样设定α=1进行LASSO估计。
分析结果鉴定出20个预测基因,并使用LASSO回归确定了它们的相关系数。这20个预测基因及其系数被用来计算mPS,该评分作为预测HGSOC患者生存的指标。
3.基于mPS的生存分析
本研究根据20个预测基因的表达水平精心制定了mPS或风险评分,并根据中位数将评分分为高风险组和低风险组,作为后续分析的基础。
为了验证 mPS 的稳健性,我们采用了双重方法。首先,使用包含 491 个样本的训练数据集,然后将 mPS 应用于剩余的 491 个样本作为测试数据集。此验证过程的结果令人信服,揭示了已确定的高风险组和低风险组之间的生存曲线存在显著差异(图 4)。训练数据集和测试数据集的对数秩 p 值均小于 0.0001,这进一步证实了 mPS 的预后功效。
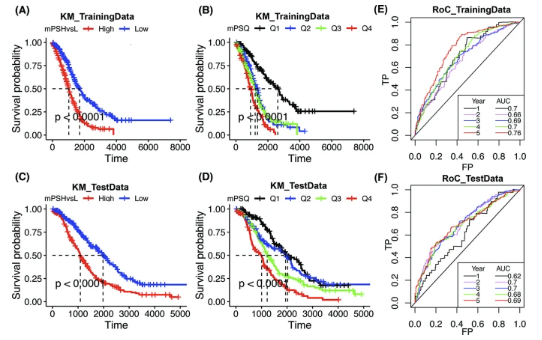
生存分析进一步揭示了显著的发现。在训练数据集中,高风险组的中位总生存(OS)时间为1024天(95% CI:914–1168),而低风险组的中位OS时间为1699天(95% CI:1446–2012)(图4A)。类似地,在测试数据集中,高风险组的中位OS时间为1091天(95% CI:1006–1234),而低风险组的中位OS时间更为有利,为1976天(95% CI:1764–2279)(图4C)。此外,对训练样本进行了细致的探索,根据mPS将其分为四分位数(Q1、Q2、Q3和Q4),其中Q4具有最高的mPS得分,而Q1具有最低的mPS得分。然后为这些等分的四分位数(Q1、Q2、Q3和Q4)生成生存曲线,以获得中位OS时间。Q1、Q2、Q3和Q4亚组的中位OS时间分别为2621、1354、1203和914天(图4B)。还获得了各自四分位数的中位mPS,并计算了四分位数的中位mPS与相应中位OS时间之间的皮尔逊相关性。在训练数据中,观察到mPS与中位OS时间之间的负相关性(r²=-0.902, p=0.049, 皮尔逊相关性)。类似地,在测试数据中也观察到mPS得分与OS时间之间的强负相关性(r²=-0.954, p=0.02)。这表明mPS评分不仅可以定性地指示生存时间,还可以定量地测量或预测生存时间(图4B和D)。
4.根据20个基因特征获得的风险评分进行预测
20基因特征是基于图3中绘制的值,通过在包含不同数据集中HGSOC样本的训练和测试数据集上进行10倍交叉验证得出的。基于这20个基因得出的mPS进一步应用于生存数据的敏感性和特异性研究,使用接收者操作特征(RoC)曲线。RoC曲线下面积(AUC)值表明了预后模型的预测能力。我们用于预测卵巢癌OS的预后模型效率较高,训练(图4E)和测试HGSOC样本(图4F)的AUC值分别约为0.70(±0.03)和0.68(±0.03),这表明我们的模型是一个非常有效的预测工具,用于确定HGSOC患者的风险或OS时间(图4)。
常用的临床参数包括FIGO分期、肿瘤分级、残留病变,以及年龄和种族,用于研究HGSOC的OS时间或预后。使用Cox回归对HGSOC的生存数据进行单变量分析,发现年龄、FIGO分期和最大结节残留病变表现出正相关(β系数>1;HR>1.2, p值<0.05),表明这些参数的值越高,与较差的生存或预后不良相关。进行多变量Cox回归分析,以调整其他显著参数的影响,得出调整后的风险比(HR)。这些参数的多变量Cox比例风险回归模型的森林图如图5A所示。最大结节残留病变在决定预后方面显示出显著效应(p<0.001),调整后的HR为1.3(95% CI=1.13–1.40)。这表明初次细胞减灭术后较大的残留病变与HGSOC患者生存率降低相关。有趣的是,mPS评分是最重要的参数(p值<0.001),调整后的风险比(调整至年龄、残留病变和FIGO)为6.1(95% CI=3.65–10.30)。进一步分析评估了添加年龄和最大结节残留病变等参数是否能增加mPS的额外预后价值或敏感性。发现单独使用mPS评分对HGSOC的5年OS的AUC为0.71,而最大结节残留病变的贡献为0.60。此外,将残留病变和年龄等参数添加到mPS中,对HGSOC患者的预测能力(AUC=0.72)只有微小提升(图5B)。因此,mPS在预测HGSOC的OS方面优于各种传统参数,如年龄和最大结节残留病变。总之,mPS本身可以作为预测HGSOC严重程度结果的关键预后因素。
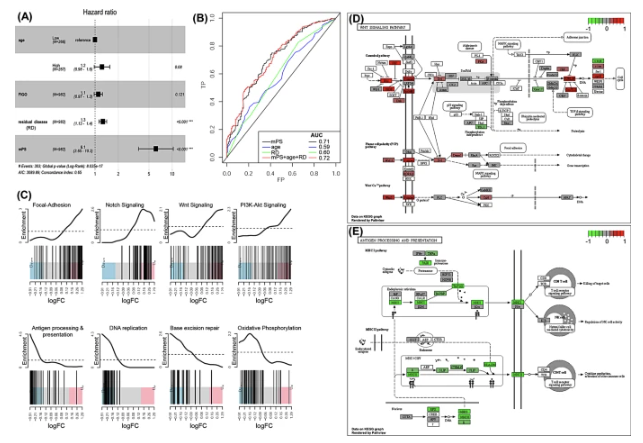
5.在高风险(预后较差)组与低风险(预后较好)组之间进行基因富集研究/通路分析
为了比较高风险(mPS较高)和低风险组之间的基因表达差异,我们进行了差异基因表达分析。与低风险组相比,我们鉴定了在高风险组中显著上调的1988个基因和显著下调的2453个基因(FDR < 0.05)。
为了探究与风险分层中这些失调基因相关的途径或事件,我们进行了基因富集研究。使用GO相关术语的基因富集分析显示,与DNA修复、呼吸电子传递链、细胞周期和DNA复制相关的途径显著下调(p < 0.05)。相反,与细胞迁移、细胞外基质相互作用、血管和血管发育相关的途径上调(p < 0.05)。使用KEGG途径数据库进行的途径分析也得出了类似结果,其中焦点粘附、Notch信号、Wnt信号、PI3-Akt信号以及调节干细胞多能性的信号途径等途径上调,而涉及抗原处理和呈递、细胞周期、DNA复制和碱基切除修复的途径下调。我们进一步研究了调控这两个途径的分子(图5D和E),发现包括Frizzled相关蛋白家族(FRP)、Wnt家族成员、色素上皮衍生因子(PEDF)、丝氨酸蛋白酶抑制剂家族F成员1(SERPINF1)、Frizzled(FZD)蛋白、BMP和活化素膜结合抑制剂(BAMBI)、段极性蛋白dishevelled(Dvl)、蛋白激酶A(PKA)、β-catenin以及转录因子样(TCF)/淋巴增强子结合因子(LEF)在内的几个关键分子在HGSOC的高风险组中上调。这种典型Wnt信号的激活可能导致细胞运动和增殖增加(图5D)。
考虑到免疫反应亚型在卵巢癌中的有利预后,我们分析了参与抗原处理和呈递信号的分子。该途径在高风险HGSOC患者中下调(p < 1.27E-10)。在这一途径中,几乎有37个分子显著下调(FDR < 0.05),包括关键介质如IFN-γ、TNF-α、免疫蛋白酶体激活因子PA28、TAP1/2、TAPBP、MHC-I(HLA-A、HLA-B、HLA-C),影响MHC-I途径介导的癌细胞杀伤。通过HLA-DMA、HLA-DMB、HLA-DOA、CLIP(CD74)、cathepsin S(CTSS)的MHC-II途径也下调,导致抗肿瘤细胞因子产生减少和其他免疫细胞激活降低。因此,根据我们的发现,免疫逃逸与高风险HGSOC患者组相关。
Sarkar, S., Saha, S.A., Swarnakar, A. et al. The molecular prognostic score, a classifier for risk stratification of high-grade serous ovarian cancer. J Ovarian Res 17, 159 (2024). https://doi.org/10.1186/s13048-024-01482-5.
声明:本材料由阿斯利康支持,仅供医疗卫生专业人士参考
审批编号:CN-148266
有效期至:2025年5月31日
排版编辑:肿瘤资讯-展思懿











 苏公网安备32059002004080号
苏公网安备32059002004080号


