卵巢癌是女性第八大常见癌症,5 年生存率仅为 30-50%。其中,I 期肿瘤生存率接近 90%,而 IV 期肿瘤生存率仅为 20%。目前鉴别卵巢良恶性肿瘤的生物标志物灵敏度低,特异性差,迫切需要新型生物标志物将有症状的女性的良性和恶性肿瘤区分开来,从而减少不必要的诊断性手术,进一步降低对生育力和医源性更年期的负面影响。近期,一项发表于Scientific Reports上的研究使用 Explore PEA 技术对 2943 种血浆蛋白进行了大规模分析,以寻找新的生物标志物,结果证实了使用高通量精确蛋白质组学来识别用于卵巢癌检测的新型血浆蛋白生物标志物的能力。在此,【肿瘤资讯】将文章整理如下。
研究背景
卵巢癌的五年生存率从I期发现时的90%下降至IV期的20%。这一变化部分反映了更具侵袭性的亚型往往在晚期被诊断出来,但同时也有观点认为早期发现可以提高生存率。卵巢癌目前是女性中第八常见的癌症,并且每年在全球范围内导致超过20万人死亡。目前,卵巢癌的发现主要依赖于症状的出现,能在I期或II期被发现的病例不足三分之一。
深入掌握卵巢癌的病因学对于确定与疾病发展过程相匹配的最佳筛查周期至关重要,然而,卵巢癌的前驱病变状态尚未被明确界定。以高级别浆液性癌为例,其假定的前驱病变状态浆液性输卵管上皮内癌(STIC)可能在首次发生易感基因突变后,经历数十年的缓慢发展过程。来自患者样本的最新分子学研究显示,STIC向卵巢癌的转变可能在大约6至7年的相对较短时间内发生。基于肿瘤尺寸和生长速率的计算预测,卵巢癌可能在原位或I期和II期存在超过4年,然后最终发展为III期和IV期。
目前,尚不存在足够准确的分子检测方法来支持基于人群的卵巢癌筛查。英国卵巢癌筛查协作试验(UKCTOCS)是规模最大的前瞻性卵巢癌筛查研究,该研究评估了针对绝经后女性的多模式筛查策略。该策略基于MUCIN-16(CA125)的分子水平升高,随后通过阴道超声检查进行确认。MUCIN-16自1983年首次作为卵巢癌的指标以来,至今仍是用于绝经后女性诊断及治疗管理的最佳单一生物标志物。多模式方法的一个难题是MUCIN-16的灵敏度相对较低,导致许多癌症被遗漏。通常,通过分析额外的生物标志物或使用阴道超声(TVU)可以取得高特异性,助于减少假阳性。目前,有盆腔症状的女性通常使用现有的分子生物标志物分析、TVU或计算机断层扫描进行检查,并以手术作为最终的诊断工具。在瑞典,接受附件肿瘤手术的女性中,接近五分之四被诊断为良性囊肿,而非癌症,需要准确的生物标志物来准确分流有症状的女性,减少不必要的诊断性手术。MUCIN-16作为单一生物标志物对早期癌症的灵敏度低,在年轻女性的许多良性妇科状况(如感染、妊娠或子宫内膜异位症)中导致高假阳性率。MUCIN-16与额外生物标志物如乳清酸性蛋白4 -二硫键核心结构域2 /人附睾蛋白4(WFDC2 / HE4)的组合,如在卵巢癌风险预测模型(ROMA)指数中使用,可以提高准确性。ROMA指数根据不同的绝经状态以不同的方式计算,最初在绝经前女性的特异性为0.75时报告的敏感性为0.77,而在绝经后女性的特异性为0.75时敏感性可达0.92。最近的荟萃分析显示,ROMA指数在绝经前和绝经后女性的总体敏感性在0.88至0.93之间,特异性在0.89至0.94之间。除了在ROMA指数中使用的MUCIN-16和WFDC2外,一些研究还指出其他蛋白质生物标志物可能对卵巢癌的分流或早期诊断具有指导意义。OVA1测试结合了五种蛋白质,并将女性分为高、中或低卵巢癌风险类别,最近在一项多中心研究中进行了评估,其中根据OVA1测试预测为低风险的个体在12个月的随访期间保持良性的比例高于仅使用CA125的预测。在最近的一项研究中,我们展示了从超过1450种血浆蛋白中选出的4至7种蛋白质生物标志物的组合在有症状女性的卵巢癌检测中优于单独的MUCIN-16。一套7种蛋白质在特异性为0.96时的灵敏度达到0.91,能够将I期和II期的良性肿瘤与恶性肿瘤区分开来。这种高准确性随后在独立队列中得到了验证。值得注意的是,我们的数据驱动方法选择生物标志物时并未包括MUCIN-16在内的7种蛋白质,这表明在没有预先假设的情况下广泛筛选蛋白质生物标志物候选物的方法具有潜力,或可远远胜于目前分子检测中早期发现卵巢癌的黄金标准。
研究方法
1.样本
研究收集了来自瑞典乌普萨拉大学Uppsala Biobank的U-CAN样本和瑞典哥德堡西部医疗区Biobankvast.se的妇科肿瘤生物银行的女性良性和恶性卵巢肿瘤的血浆样本,并纳入了因怀疑卵巢癌而行手术治疗,最终手术诊断结果显示为良性病变的患者的样本。排除标准包括接受新辅助化疗的患者或病理上确定为来自其他组织的转移性肿瘤。样本收集遵循所有当地指南和法规,U-CAN样本收集时间为2012年至2018年,哥德堡样本收集时间为2016年至2018年。肿瘤由专门从事妇科癌症的病理学家根据国际妇产科联盟(FIGO)标准进行组织学、分级和分期检查。两个队列包含混合肿瘤组织学。U-CAN样本中,60.1%为高级别浆液性(HGS),8.7%为低级别浆液性(LGS),其余为其他类型。哥德堡样本中,70.6%为HGS,8.2%为LGS,7.0%为粘液性,其余为其他类型。研究得到了乌普萨拉和哥德堡地区伦理委员会的批准,并遵循赫尔辛基宣言指南获得了所有参与者的知情书面同意。
2.蛋白质组学
使用PEA Explore153619测定法和PEA Explore3072扩展测定法分析样本。
3.数据分析和统计
所有计算均采用R软件完成。单变量比较通过单侧Wilcoxon秩和检验进行,随后使用Holm校正法对p值进行调整。多变量分析首先利用"caret包"中的递归特征选择方法进行特征筛选,随后构建朴素贝叶斯模型,并采用四折交叉验证法来优化模型参数。模型的性能通过ROC曲线在训练队列和验证队列中分别进行评估,并使用AUC统计量和DeLong测试来比较两组队列的性能。相关性分析则采用Spearman相关系数计算方法。所有统计分析均按照相关指南和规定进行。
研究结果
1.用于早期检测的多重单一生物标志物
本研究对两个独立临床队列共571个样本进行了分析,这些样本来自怀疑患有卵巢癌的女性,她们最终被诊断为良性或恶性肿瘤。样本通过Explore19 PEA18进行了检测。第一组样本(表1,发现队列)来自瑞典哥德堡西部医疗区的队列,第二组(表1,复制队列)来自瑞典乌普萨拉大学Uppsala Biobank的U-CAN。
其中443个样本采用了Olink Explore3072扩展检测法。部分样本已经使用Olink Explore1536检测法进行了检测,相关结果已在文献15,20中发表。两个检测板(1536和3072)均包含1536个检测项目。在发现队列中有107个样本,在复制队列中有163个样本具有这两次检测的完整数据。在之前的研究15中,哥德堡队列作为发现队列,乌普萨拉队列用于复制,本研究沿用了这一分组方式。我们首先分别分析了两种PEA Explore检测法的数据,以增强单变量分析的统计功效。这包括来自发现和复制队列的111个和237个样本分别进行了Explore1536检测,以及167个和276个样本分别进行了Explore3072检测。单变量分析通过三种方式执行:良性与早期卵巢癌(I期和II期)、良性与晚期(III期和IV期)以及良性与任何阶段(I-IV期)的对比。在发现队列中经过多重比较校正后,共有4种蛋白质在良性与早期癌症间显著不同(图1A),有163种在良性与晚期间(图1B),最终有129种在良性与任何阶段间(图1C)显著不同。这些296种蛋白质中,有几种在多个比较类别中显著不同,累计涉及171种独特蛋白质。
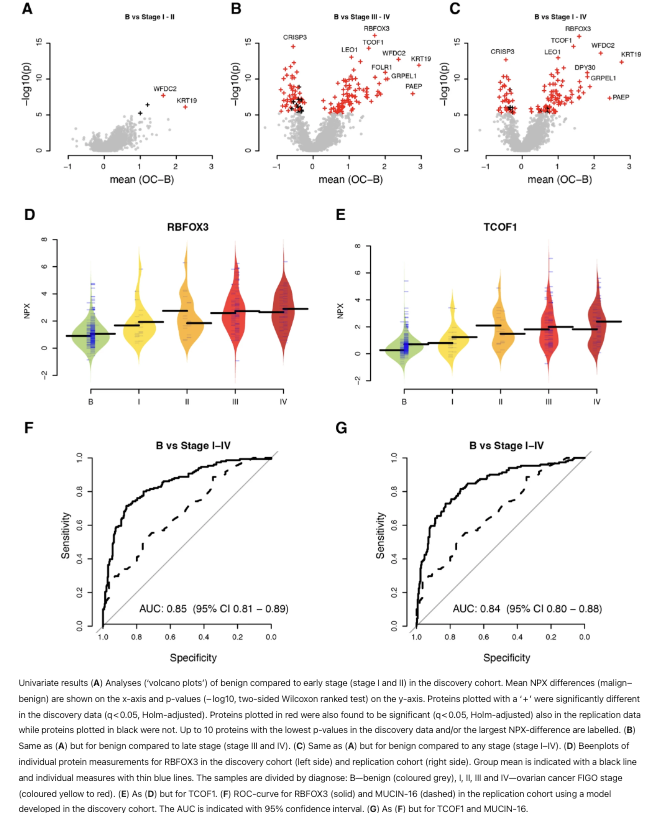
随后在复制队列中分析了这296个关联,发现所有296个关联在复制队列中变化趋势一致,且279个(94.3%)具有名义上的显著性差异。在复制队列中经过多重比较校正后,269个(90.9%)的关联仍然显著。所有单变量结果均列在补充表1中。MUCIN-16作为具有复制性能的生物标志物之一,在发现数据中,比较良性与任何阶段癌症时,按p值排序为第139个最显著的关联。RBFOX3和TCOF1是两种在比较良性与任何阶段癌症时p值最低的蛋白质,它们在两个队列中按阶段获得的NPX值分布见图1D和E。接下来,我们比较了这两种生物标志物作为单一价值分类器区分良性与任何阶段的性能。在复制队列中,RBFOX3的AUC为0.85(95%置信区间0.81-0.89,图1F),TCOF1的AUC为0.84(0.80-0.88),而MUCIN-16的AUC为0.68(0.61-0.75)。在图F和G中,选定的生物标志物显示为实线,而MUCIN-16(用PEA测量)的性能以虚线表示。这两种蛋白质的AUC均显著高于MUCIN-16(所有p值<1.1×10-4,DeLong测试)。两个队列的临床CA125测量值与PEA等效物的相关性为中等(Spearman’s’ Rho = 0.53),但具有显著性相关性(p < 2.8×10-9)。两个队列中,CA125测量值在区分良性与早期(I和II期)、晚期(III和IV期)和任何阶段(I-IV期)肿瘤方面的AUC分别为0.70(0.56—0.85)、0.93(0.87–0.98)和0.87(0.80—0.94)。在临床上常用的35 U/ml截断值下,仅CA125在区分上述三类情况时分别实现了0.78(0.61–0.94)、1.00(1.00–1.00)和0.94(0.89–0.99)的敏感性,以及0.44(0.31–0.59)、0.44(0.28–0.59)和0.44(0.28–0.59)的特异性。
2. 联合生物标志物增强诊断精度
我们进一步构建了区分以下几组的多变量预测模型:(i) 良性与早期肿瘤(I期和II期),(ii) 良性与晚期肿瘤(III期和IV期),以及 (iii) 良性与任何阶段肿瘤(I-IV期)。在所有三个模型中,我们首先使用发现队列中所有蛋白质的特征选择方法。然后,所选蛋白质被用来构建并优化一个风险评分分类器,其评分范围为0至1。我们根据风险评分设定了一个预测恶性的截断值,旨在实现至少95%的敏感性。接着,我们将该模型应用于复制队列,使用相同的截断值计算风险评分,并通过这些评分来评估模型的最终性能。在这项分析中,两个PEA检测(Explore 1536和3072扩展,方法部分有详细说明)的数据被合并,仅保留了无缺失值的蛋白质和个体。最终数据集包含2934个蛋白质,这些蛋白质在两个队列中均有测量结果,其中发现队列有107个样本,复制队列有163个样本。
区分早期与良性的模型由WFDC2、FOLR1和KRT19三种蛋白组成,在复制队列中AUC达到了0.90(图2A),与发现队列的表现(AUC=0.94,p=0.45,DeLong方法,表2)没有统计学差异,表明模型具有良好的预测稳定性。
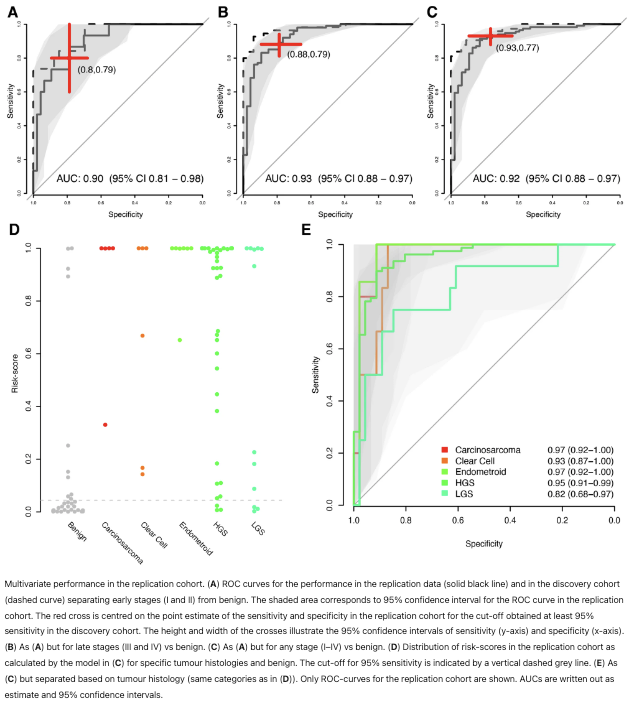 图2
图2
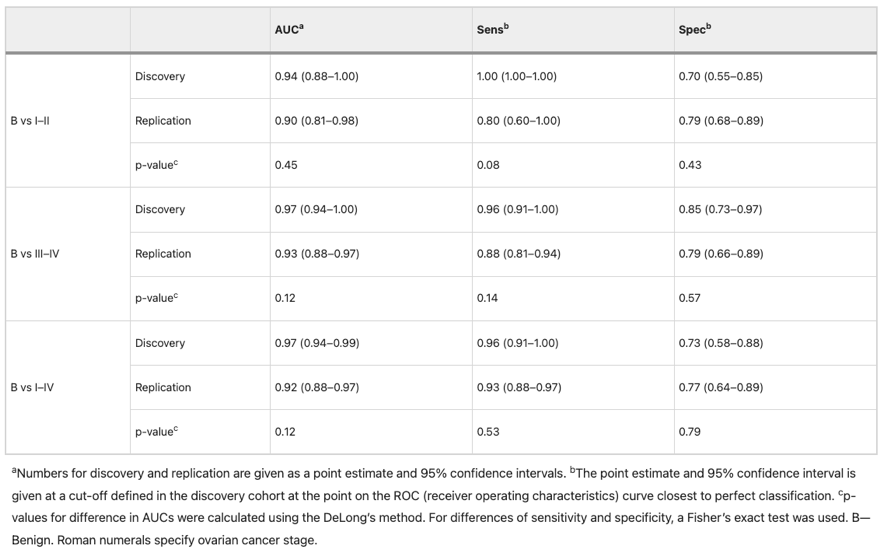
与单独使用CA125相比,该三蛋白模型在区分早期癌症与良性病变方面显著提高了AUC(p<0.023,DeLong方法)。晚期与良性比较的模型选用了RBFOX3和WDFC2两种蛋白,复制队列中的AUC为0.93(图2B),与发现队列的0.97相比(p>0.12,DeLong方法,表2),性能相当。最后,区分良性与任何阶段的模型也由WFDC2、FOLR1和KRT19三种蛋白组成,在复制队列中的AUC为0.92(图2C),与发现队列的0.97相比(p>0.12,DeLong方法,表2),同样显示出良好的一致性。尽管多变量蛋白模型在区分晚期(III和IV)及任何阶段(I-IV)与良性相比时AUC值一贯高于单独使用CA125,但这些差异在统计上不显著(p>0.19,DeLong方法)。此外,使用发现队列我们还为每个模型开发了一个至少需要95%敏感性的截断值以区分恶性与良性,随后也应用于复制队列。对于所有三个模型,在复制数据中得到的敏感性和特异性的点估计值没有统计学差异(所有p值均>0.08,Fisher精确检验,表2)。总体而言,这里为早期、晚期和任何阶段与良性条件生成的三个模型总共只使用了四种蛋白(WFDC2、FOLR1、KRT19和RBFOX3)。我们还比较了良性与任何阶段模型在不同肿瘤组织学中的性能(图2D)。在比较不同组织学之间的原始风险评分时,我们发现肉瘤、内膜样和高级别浆液性(HGS)与低级别浆液性(LGS)之间存在名义上显著的差异(p值分别为0.037、0.022和0.0021),LGS组的评分较低(图2D)。经过多重假设检验调整后,只有HGS与LGS之间的差异仍然显著(q=0.021)。我们还比较了不同组织学中预测为假阴性的样本比例,并发现LGS相较于HGS有名义上显著更高比例的假阴性(p=0.029,Fisher精确检验),尽管这一显著性在多重假设检验调整后不复存在(q=0.29)。最后,我们比较了模型在区分良性与特定组织学中的预测性能(图2E),尽管在区分LGS与良性相比所有其他比较中存在性能较差的趋势,我们发现在任何类别中估计的AUC之间没有统计学差异(所有名义p值均>0.063,DeLong方法)。
Ivansson, E., Hedlund Lindberg, J., Stålberg, K. et al. Large-scale proteomics reveals precise biomarkers for detection of ovarian cancer in symptomatic women. Sci Rep 14, 17288 (2024). https://doi.org/10.1038/s41598-024-68249-2
排版编辑:肿瘤资讯-展思懿











 苏公网安备32059002004080号
苏公网安备32059002004080号


